GPB |深圳市人民医院/中科院计算所团队发表肿瘤免疫单细胞长非编码RNA图谱研究
T细胞检查点抑制疗法已在许多癌症中显示出显著的临床获益,但不同癌症患者治疗效果存在较大差异,这主要归因于当前对治疗应答或耐药性的机制尚不清楚。越来越多的证据表明,长非编码RNA(lncRNAs)在免疫细胞中广泛表达,并通过调节T细胞分化和功能在癌症免疫中发挥重要作用。目前,大多数肿瘤免疫相关的单细胞测序(scRNA-seq)研究普遍集中于编码基因,忽略了大量的lncRNA。lncRNA的表达相对较低且具有细胞特异性,在单细胞水平对lncRNA进行分析仍具有挑战性。
近日,深圳市人民医院李富荣、中国科学院计算技术研究所赵屹团队在Genomics, Proteomics & Bioinformatics期刊(IF=7.691,1区)发表了题为“Single-cell Long Non-coding RNA Landscape of T Cells in Human Cancer Immunity”的研究文章。研究团队利用来自3种癌症类型不同组织的20,000多个T细胞单细胞全长转录组数据,创建了一个完整的T细胞lncRNA数据集,并分析了与不同T细胞状态相关的功能,为探索lncRNA在肿瘤T细胞中的调控机制提供了丰富的数据资源。

为了研究不同患者、组织和癌症类型T细胞中lncRNA图谱,研究团队利用已公开发表的Smart-seq2全长单细胞RNA测序数据,共计24,068个T细胞测序数据。其中9,878个细胞来自结直肠癌(CRC)患者、10,188个细胞来自非小细胞肺癌(NSCLC)患者、4,002个细胞来自肝细胞癌(HCC)患者。这些T细胞收集自每个患者的外周血、邻近正常组织和肿瘤组织。
首先,研究人员使用从头组装的方法,评估单细胞层面的转录本组装情况。基于HCC患者的T细胞数据集,研究人员分析了每个T细胞的组装程度,发现平均有4,752个转录本可在单细胞水平上被组装出来,平均仅69.8%与已知基因相匹配,该结果暗示有30%左右的基因是单细胞层面发现的潜在的新基因 (图1)。接下来,研究团队分析比较了三种不同层面组装方法(细胞水平、细胞类型水平、组织水平)的结果,发现基于细胞类型水平策略的组装转录本数量明显高于细胞水平和组织水平,并且来自细胞类型水平的匹配转录本的平均数量是bulk-seq方法的两倍多。
基于细胞类型层面的拼接策略,将来自三种癌症类型所组装的转录本合并,共获得了751,710个初始基因集合,经过一系列严格的筛选及过滤条件,最终获得了9,433个新的lncRNA基因,并对部分基因进行了Sanger测序的实验验证,该集合已上传到NONCODE数据库,供免费下载。 同时,研究发现新的lncRNAs比已知lncRNAs在肿瘤T细胞中显示出更高、更广泛的表达水平。
为了进一步探索在肿瘤免疫中与T细胞状态相关的特征lncRNAs,研究团队利用MetaCell方法将scRNA-seq数据集从单细胞转变为metacell,以避免单细胞水平所带来的对低表达基因分析的扰动 (图2)。基于metacell图谱,该研究共鉴定了154个与T细胞耗竭、活性以及调节性显著相关的lncRNAs,并对这些基因的特征进行了系统地分析。
为进一步解析免疫调控相关lncRNA的功能机制,研究团队构建了由编码基因及lncRNA基因所组成的共表达网络,并对84个特征lncRNA的功能进行了注释,功能分析结果与T细胞的状态相吻合。例如,耗竭相关的特征lncRNAs显著富集免疫抑制功能,包括免疫应答的负调控、T细胞活化的负调控、IL-10生物合成过程的正调控等。
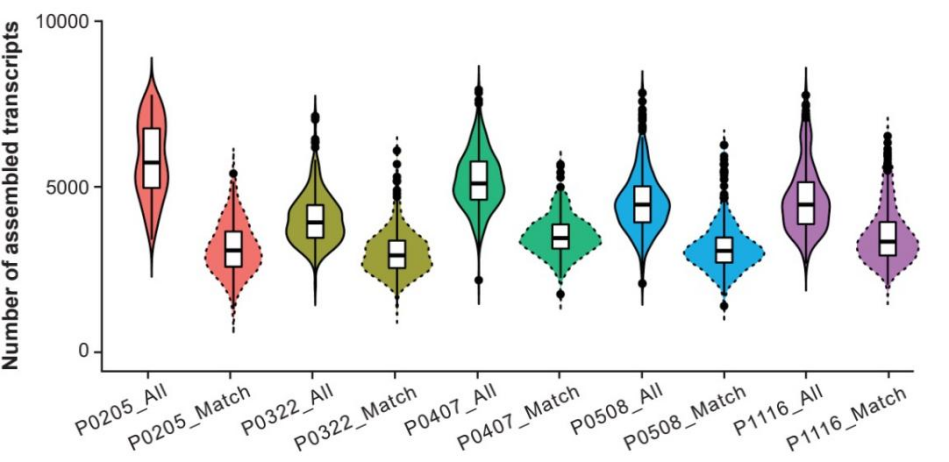
图1. HCC患者的组装转录本数量。来源:GPB

图2. Metacell图谱。来源:GPB
综上所述,该研究基于2万余例全长单细胞转录组测序数据,通过对长非编码RNA的基因鉴定、表达模式分析、共表达网络分析等,系统研究了与癌症免疫调控相关的长非编码RNA基因,并对其生物学机制进行初步探究。虽然具体机制还需进一步用实验验证,但该工作为研究肿瘤免疫中T细胞调控机制及肿瘤免疫生物标志物的开发提供了一个新的视角和数据资源。
参考文献:
Haitao Luo, Dechao Bu, Lijuan Shao, Yang Li, Liang Sun, Ce Wang, Jing Wang, Wei Yang, Xiaofei Yang, Jun Dong, Yi Zhao, Furong Li. bioRxiv 2020.07.22.215855; doi: https://doi.org/10.1101/2020.07.22.215855
【收藏此页】 【打印本页】 【关闭窗口】
 官方微信
官方微信




